Una colaboración pionera en ‘conectómica’ ha reconstruido con éxito un milímetro cúbico de tejido cerebral, pero los investigadores todavía están arañando la superficie de su complejidad.
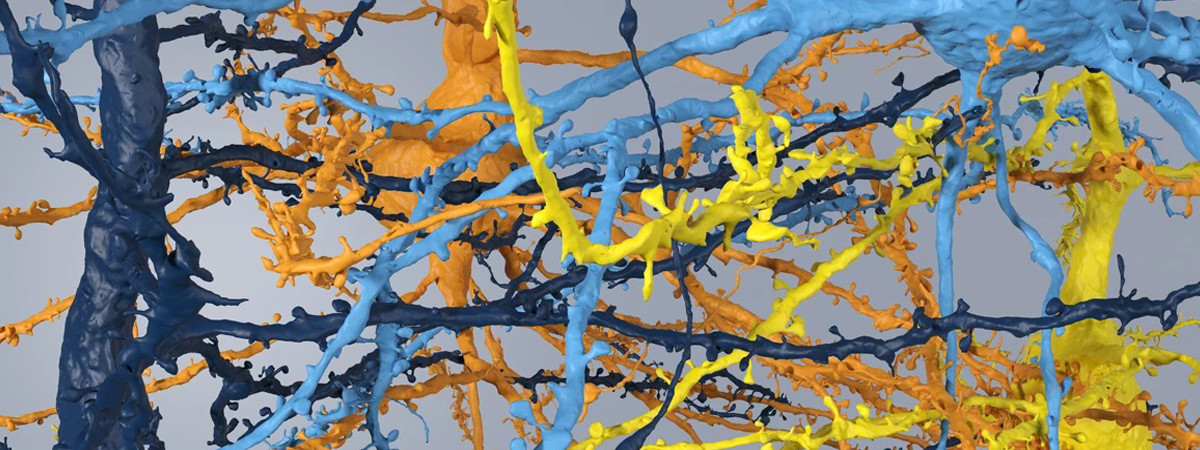
Procesos neuronales de cuatro neuronas de distintos colores mapeados por el proyecto Machine Intelligence from Cortical Networks. Crédito: Explorador de MICrONS
Un milímetro cúbico es un volumen diminuto, menos que una lágrima. Pero un milímetro cúbico de cerebro de ratón está densamente poblado de decenas de miles de neuronas y otras células en un tejido arquitectónico asombrosamente complejo.
Reconstruir disposiciones tan elaboradas requiere un esfuerzo monumental, pero los investigadores afiliados al programa Machine Intelligence from Cortical Networks (MICrONS) lo lograron. Se necesitaron 100 millones de dólares y años de esfuerzo de más de 100 científicos, coordinados por 3 grupos que nunca habían colaborado. Hubo semanas de trasnoches y un minucioso esfuerzo global de revisión que continúa incluso ahora, para un volumen que representa sólo el 0,2% del cerebro típico de un ratón. A pesar de los obstáculos, el núcleo del proyecto, concebido y financiado por la Actividad de Proyectos de Investigación Avanzada de Inteligencia de Estados Unidos (IARPA), está completo.
El paquete resultante incluye una reconstrucción por microscopía electrónica 3D de alta resolución de las células y orgánulos en dos volúmenes separados de la corteza visual del ratón, junto con imágenes fluorescentes de la actividad neuronal de los mismos volúmenes. Incluso los coordinadores del proyecto MICrONS, que describen la reunión del consorcio por parte de IARPA como una “boda forzosa” de esfuerzos de investigación paralelos, quedaron gratamente sorprendidos por el resultado. “Se formó este equipo contiguo y hemos estado trabajando muy bien juntos, es impresionante”, dice Andreas Tolias, neurocientífico de sistemas que dirigió el esfuerzo de imágenes funcionales en la Facultad de Medicina Baylor en Houston, Texas.
El proyecto MICrONS es un hito en el campo de la “conectómica”, cuyo objetivo es desentrañar la organización a escala sináptica del cerebro y trazar los circuitos que coordinan las numerosas funciones del órgano. Los datos de estos dos primeros volúmenes ya están proporcionando a la comunidad de neurociencia un recurso valioso. Pero este trabajo también está llevando a los científicos a un territorio nuevo, extraño y desafiante. «La principal víctima de esta información es la comprensión», afirma Jeff Lichtman, pionero de la conectómica en la Universidad de Harvard en Cambridge, Massachusetts. «Cuanto más sabemos, más difícil es convertir esto en un modelo simple y fácil de entender de cómo funciona el cerebro».
Cortocircuitos
Hay muchas formas de observar el cerebro, pero para los investigadores de la conectividad, la microscopía electrónica ha demostrado ser especialmente poderosa.
En 1986, científicos de la Universidad de Cambridge, Reino Unido, utilizaron microscopía electrónica de cortes seriados para generar un mapa completo del sistema nervioso del nematodo Caenorhabditis elegans1 . Ese conectoma fue un logro histórico en la historia de la biología. Requirió la ardua anotación manual y la reconstrucción de unas 8.000 imágenes 2D, pero produjo la piedra de toque para comprender el sistema nervioso de este modelo animal simple pero importante.
No existe ningún recurso comparable para animales más complejos, pero las primeras incursiones en el conectoma de roedores han dado pistas de lo que dicho mapa podría revelar. Lichtman recuerda el ensamblaje que él y sus colegas produjeron en 2015 a partir de una sección de neocórtex de ratón de 1.500 micrones cúbicos, aproximadamente una millonésima parte del volumen utilizado en el proyecto MICrONS2 . “La mayoría de la gente se sorprendió al ver la densidad de cables en cualquier pequeña parte del cerebro”, dice.
De manera similar, Moritz Helmstaedter, investigador de conectómica del Instituto Max Planck para la Investigación del Cerebro en Frankfurt, Alemania, dice que los esfuerzos de su equipo 3 para reconstruir una región densamente poblada de la corteza somatosensorial del ratón, que procesa sensaciones relacionadas con el tacto, en 2019 desafiaron los dogmas existentes, especialmente la suposición de que las neuronas de la corteza están conectadas al azar. “Demostramos explícitamente que eso estaba mal”, dice Helmstaedter. «Encontramos esta precisión extrema». Estos y otros estudios han ayudado colectivamente a consolidar la importancia de los mapas de circuitos basados en microscopía electrónica como complemento de técnicas como la microscopía óptica y los métodos moleculares.
Mas grande y mejor
La motivación de IARPA para el proyecto MICrONS se basó en la inteligencia artificial. El objetivo era generar un mapa conectómico detallado a escala de milímetros cúbicos, que luego podría someterse a «ingeniería inversa» para identificar principios arquitectónicos que podrían guiar el desarrollo de redes neuronales artificiales biológicamente informadas.
Tolias, el neurocientífico Sebastian Seung de la Universidad de Princeton en Nueva Jersey y el neurobiólogo Clay Reid del Instituto Allen de Ciencias del Cerebro en Seattle, Washington, habían solicitado de forma independiente financiación para contribuir a elementos separados de este programa. Pero los funcionarios del programa de IARPA eligieron combinar los tres equipos en un solo consorcio, incluida una red más amplia de colaboradores, y emitieron $100 millones en 2016 para apoyar un esfuerzo de cinco años.
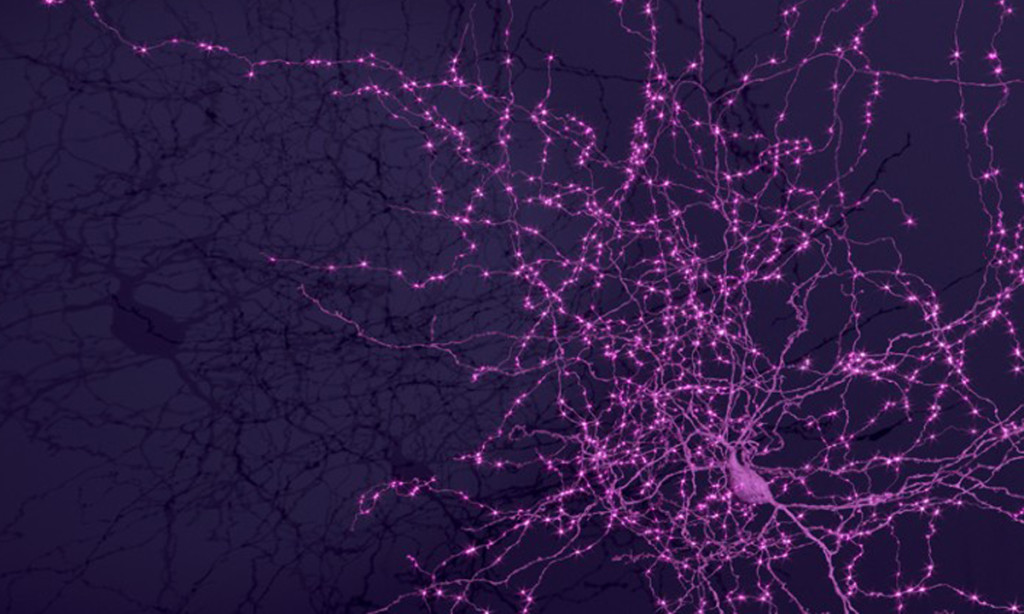
El equipo de MICrONS seleccionó dos áreas de la corteza visual del ratón: el milímetro cúbico antes mencionado y un volumen mucho más pequeño que sirvió como piloto para el flujo de trabajo. Estos fueron elegidos para que el equipo pudiera investigar las interacciones entre regiones dispares en la vía visual, explica Tolias, quien supervisó el aspecto de imágenes de actividad cerebral del trabajo en Baylor. Para lograrlo, los investigadores diseñaron genéticamente un ratón para que expresara un «gen informador» sensible al calcio, que produce una señal fluorescente cada vez que una neurona o una población de neuronas se activa. Luego, su equipo reunió imágenes de vídeo de diversas escenas realistas, que el animal observó con cada ojo de forma independiente durante dos horas mientras un microscopio rastreaba la actividad neuronal.
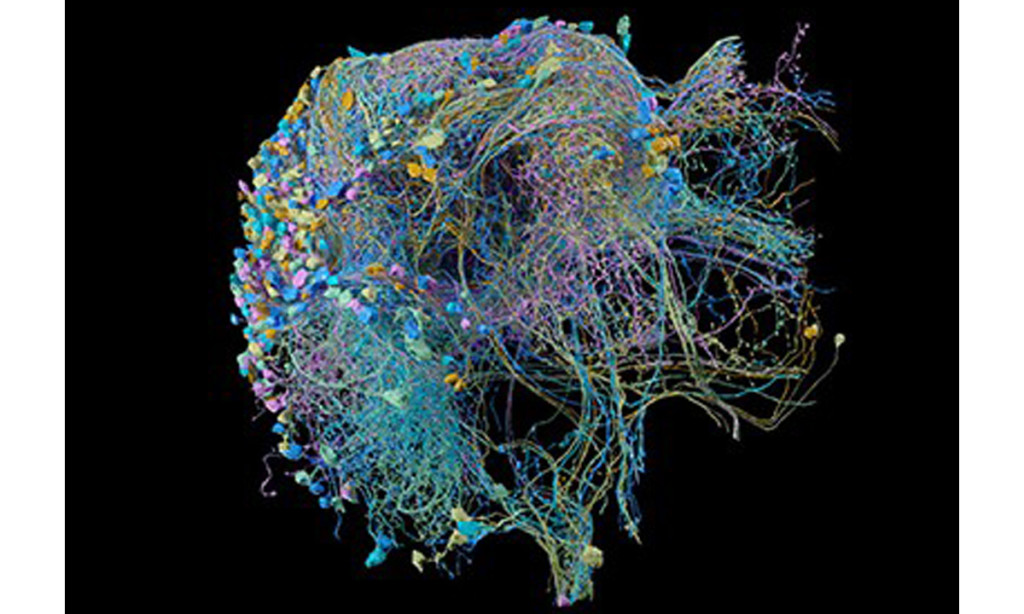
Luego, el ratón fue enviado a Seattle para su preparación y obtención de imágenes de los volúmenes cerebrales relevantes, y la presión aumentó otro nivel. Nuno da Costa, neuroanatomista e investigador asociado del Instituto Allen, dice que él y Tolias comprimieron los horarios de sus grupos para acomodar la etapa final, que requiere mucho tiempo, de reconstrucción y análisis digital realizada por el grupo de Seung. “Realmente nos esforzamos para cumplir: fallar lo antes posible para poder corregir el rumbo a tiempo”, dice da Costa. Esto significó una carrera contrarreloj para extirpar el tejido, tallarlo en rodajas ultrafinas y luego obtener imágenes de las rodajas teñidas con una flota de cinco microscopios electrónicos. «Invertimos en este enfoque en el que podíamos comprar máquinas muy antiguas y realmente automatizarlas para hacerlas súper rápidas», dice da Costa. De este modo, los investigadores pudieron maximizar el rendimiento y contaron con copias de seguridad en caso de que fallara un microscopio.
Para la primera fase del proyecto, que implicó la reconstrucción del volumen cortical más pequeño, la sección del tejido se redujo a los heroicos esfuerzos de Agnes Bodor, neurocientífica del Instituto Allen, que pasó más de un mes recolectando manualmente varios miles de tubos de 40 nanómetros. -secciones gruesas de tejido utilizando un instrumento con hoja de diamante conocido como microtomo, dice da Costa. Ese esfuerzo manual era insostenible para el volumen mayor de la fase dos del proyecto, por lo que el equipo de Allen adoptó un enfoque automatizado. Durante 12 días de trabajo supervisado las 24 horas del día, el equipo generó casi 28.000 secciones que contenían más de 200.000 celdas 4 . Se necesitaron seis meses para obtener imágenes de todas esas secciones, lo que arrojó unos 2 petabytes de datos.
Los equipos de Allen y Baylor también colaboraron para vincular las células con imágenes fluorescentes con sus contrapartes en el volumen conectómico reconstruido.

A lo largo de este proceso, el equipo de Allen transmitió sus conjuntos de datos al equipo de la Universidad de Princeton. La microscopía electrónica de cortes en serie es una técnica bien establecida, pero el ensamblaje del volumen reconstruido implica un trabajo computacional considerable. Las imágenes deben alinearse con precisión entre sí teniendo en cuenta cualquier deformación asociada a la preparación o la imagen, y luego se someten a «segmentación» para identificar y anotar neuronas, células no neuronales como la glía, orgánulos y otras estructuras. «La tecnología revolucionaria de MICrONS fue la alineación de imágenes», dice Seung. Esta parte es crucial, porque un paso en falso en la colocación de un solo corte puede descarrilar el resto del proceso de reconstrucción. La curación manual sería totalmente impráctica en la escala de milímetros cúbicos. Pero a través de su trabajo en la fase uno, el equipo desarrolló un flujo de trabajo de reconstrucción que podría ampliarse para un volumen cerebral más grande, y los avances continuos en los métodos de aprendizaje profundo hicieron posible automatizar los pasos clave de alineación.
Para comprobar el trabajo, Sven Dorkenwald, que era estudiante de posgrado en el laboratorio de Seung y ahora es investigador en el Instituto Allen, desarrolló un marco de revisión para refinar las reconstrucciones del equipo y garantizar su fidelidad biológica. Este enfoque, que verificó las rutas de los procesos neuronales a través del conectoma, talló los volúmenes en ‘supervóxeles’ (formas tridimensionales que definen características celulares o subcelulares segmentadas, que pueden reorganizarse para mejorar la precisión conectómica) y Dorkenwald dice que el conjunto de datos final de MICrONS tenía 112 mil millones de ellos. El sistema es análogo a la enciclopedia en línea Wikipedia en algunos aspectos, lo que permite a muchos usuarios contribuir con ediciones en paralelo y al mismo tiempo registrar el historial de cambios. Pero incluso la revisión colaborativa es lenta: Dorkenwald estima que cada axón (las proyecciones neuronales que transmiten señales a otras células) en el conjunto de datos MICrONS tarda hasta 50 horas en corregirse.
Trazando un nuevo territorio
El equipo MICrONS publicó un resumen 5 de los resultados de la fase uno en 2022. Muchos de sus otros hallazgos iniciales aún esperan publicación, incluida una descripción detallada del trabajo de la fase dos, aunque actualmente está disponible como artículo preimpreso 4 . Pero ya existen algunas demostraciones importantes de lo que la conectómica puede ofrecer a esta escala.
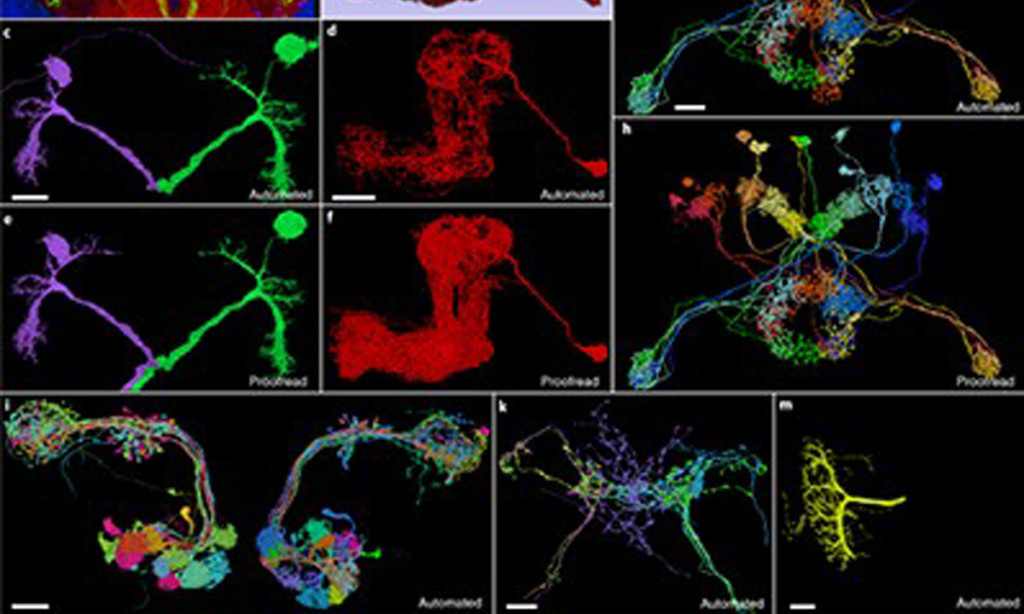
Una preimpresión de MICrONS, por ejemplo, describe lo que quizás sea el mapa de circuito más completo hasta el momento para una columna cortical 6 , una disposición en capas de neuronas que se cree que es la unidad organizativa fundamental de la corteza cerebral. La reconstrucción del equipo arrojó un censo detallado de todos los diferentes tipos de células que residen en la columna y reveló patrones previamente desconocidos sobre cómo se conectan entre sí los distintos subtipos de neuronas. «Las células inhibidoras tienen esta notable especificidad hacia algunos tipos de células excitadoras, incluso cuando estas células excitadoras están mezcladas en la misma capa», dice da Costa. Estos conocimientos podrían conducir a una clasificación más precisa de las células que estimulan o suprimen la actividad de los circuitos y revelar las reglas subyacentes que guían el cableado de esos circuitos.
Lo más importante, dice Tolias, es que el proyecto MICrONS iba más allá del conectoma: “Se trataba de imágenes funcionales a gran escala del mismo ratón”. Gran parte del trabajo de su equipo se ha centrado en traducir las mediciones de actividad basadas en indicadores de calcio en modelos computacionales de próxima generación. En 2023, los investigadores publicaron una preimpresión que describe la creación de un «gemelo digital» basado en el aprendizaje profundo a partir de respuestas corticales medidas experimentalmente a estímulos visuales 7 . Las predicciones generadas por este «gemelo» pueden luego probarse, refinando aún más el modelo y mejorando su precisión.
Un producto sorprendente y valioso del esfuerzo de MICrONS son las moscas de la fruta. Al principio del proyecto, el equipo de Seung comenzó a explorar datos de microscopía electrónica de secciones en serie del cerebro de Drosophila melanogaster producidos por investigadores del Campus de Investigación Janelia del Instituto Médico Howard Hughes en Ashburn, Virginia 8 . «Me di cuenta de que, gracias a que habíamos desarrollado esta tecnología de alineación de imágenes, teníamos la oportunidad de hacer algo que la gente pensaba que era imposible», dice Seung. Su equipo, incluido Dorkenwald, utilizó los datos de Janelia como campo de pruebas para los algoritmos que se habían desarrollado para MICrONS. El resultado fue el primer ensamblaje completo del conectoma cerebral de la mosca de la fruta: alrededor de 130.000 neuronas en total 9 .
Dado que el cableado del sistema nervioso generalmente se conserva en las moscas de la fruta, Dorkenwald está entusiasmado con la forma en que estos datos, a los que se puede acceder públicamente en http://flywire.ai , podrían permitir experimentos futuros. «Se pueden obtener imágenes funcionales en una mosca y, como se pueden encontrar las mismas neuronas en el conectoma, se podrán realizar estos análisis de la estructura funcional», afirma.
El conectoma del ratón no será tan sencillo, porque la conectividad varía de un individuo a otro. Sin embargo, los datos de MICrONS son valiosos para la comunidad de neurociencias, afirma Helmstaedter, que no formó parte del proyecto MICrONS. «Son datos fantásticos e inspiran a la gente a mirarlos y verlos», afirma. También existe el poder de demostrar lo que es posible y cómo se podría hacer mejor. «Primero tienes que hacer algo de fuerza bruta para descubrir dónde puedes hacerlo más fácil en la siguiente ronda», dice Kristen Harris, neurocientífica de la Universidad de Texas en Austin. «Y el hecho de hacerlo, simplemente hacer el trabajo, es simplemente espectacular».
Tierra desconocida
Incluso a medida que avanza el análisis del conjunto de datos MICrONS, sus limitaciones ya están quedando claras. Por un lado, se necesitarán volúmenes de otras regiones corticales distintas para identificar características que se observan ampliamente en todo el cerebro frente a aquellas características que son distintas de la corteza visual. Y muchos axones de este primer milímetro cúbico se conectarán inevitablemente a puntos desconocidos, señala Lichtman, lo que limita la capacidad de los investigadores para comprender completamente la estructura y función de los circuitos que contiene.
Ampliar la escala será aún más difícil. Lichtman estima que una reconstrucción de todo el cerebro mediante microscopía electrónica produciría aproximadamente un exabyte de datos, lo que equivale a mil millones de gigabytes y es 1.000 veces mayor que los petabytes de datos producidos por el proyecto MICrONS. «Esto puede ser un ‘disparo a Marte’; en realidad es mucho más difícil que ir a la Luna», dice.
Aún así, la carrera está en marcha. Un esfuerzo importante es BRAIN CONNECTS, un proyecto respaldado por los Institutos Nacionales de Salud de EE. UU. con una financiación de 150 millones de dólares y coordinado por varios investigadores, incluidos Seung, da Costa y Lichtman. «Aún no estamos entregando todo el cerebro del ratón, pero estamos probando si es posible», dice da Costa. «Mitigar todos los riesgos, reducir el costo y ver si realmente podemos preparar una muestra de todo el cerebro de un ratón o de todo el hemisferio».
Paralelamente, Lichtman está trabajando con un equipo de Google Research en Mountain View, California, dirigido por el científico informático Viren Jain, que colaboró con MICrONS y también forma parte del equipo de liderazgo de BRAIN CONNECTS, para mapear volúmenes considerables de la corteza humana utilizando electrones. microscopía. Ya han publicado datos de su primer milímetro cúbico y tienen planes de comenzar a trazar otras regiones de personas con diversas afecciones neurológicas

Estos esfuerzos requerirán herramientas mejoradas. La estrategia de microscopía electrónica de secciones en serie que utilizó MICrONS requiere demasiado trabajo para usarla a escalas mayores y produce datos de calidad relativamente baja que son difíciles de analizar. Pero están surgiendo alternativas. Por ejemplo, los métodos de microscopía electrónica de «cara de bloque», en los que se visualiza la muestra como un volumen sólido y luego se elimina gradualmente con un haz de iones de alta intensidad, requieren menos trabajo en términos de alineación de la imagen y pueden aplicarse a secciones gruesas de tejido que son más fáciles de manejar. Estos métodos se pueden combinar con microscopios electrónicos de barrido de haces múltiples de última generación, que obtienen imágenes de muestras utilizando hasta 91 haces de electrones simultáneamente, acelerando así la recopilación de datos. «Ese es uno de los principales candidatos para ampliar hasta un cerebro de ratón completo», afirma Seung, que trabajará con Lichtman en esta estrategia.
Una mayor automatización y más herramientas de inteligencia artificial también serán ventajas. Helmstaedter y sus colegas han estado buscando formas de simplificar el ensamblaje de imágenes con un algoritmo de segmentación automatizado llamado RoboEM, que rastrea los procesos neuronales con una mínima intervención humana y puede eliminar potencialmente gran parte de la carga actual de revisión 11 . Aún así, la preparación de muestras y las imágenes de mayor calidad son probablemente la verdadera clave para la eficiencia a escala, afirma Helmstaedter. «Cuanto mejores sean sus datos, menos tendrá que preocuparse por la automatización».
Independientemente de cómo se generen, entender estos mapas de conectomas requerirá algo más que tecnología sofisticada. Tolias cree que «será casi imposible» replicar el acoplamiento de estructura y actividad producido por MICRONS a escala de todo el cerebro. Pero tampoco está claro si eso será necesario y en qué medida se puede inferir información funcional a través de una mejor comprensión de la estructura y organización del cerebro.
Para Lichtman, el valor del conectoma trascenderá en última instancia la ciencia convencional basada en hipótesis. Un conectoma «te obliga a ver cosas que no estabas buscando y, sin embargo, te están mirando a la cara», dice. “Creo que si hacemos un cerebro de ratón completo, habrá una cantidad infinita de ‘wow, ¿en serio?’ descubrimientos”.